Un guide étape par étape sur la façon de collecter et de prétraiter l'imagerie satellite data pour les algorithmes d'apprentissage automatique.
Cet article est la première partie d'une série en deux parties qui explore les utilisations des algorithmes d'apprentissage automatique sur l'imagerie satellitaire. Nous nous concentrerons ici sur les étapes de collecte et de prétraitement de data , tandis que la deuxième partie présentera l'utilisation de divers algorithmes d'apprentissage automatique.
TL;DR
Cet article :
Nous utiliserons la détection et la classification des champs agricoles comme exemple. Python et le notebook jupyter sont utilisés pour les étapes de prétraitement. Le notebook jupyter permettant de reproduire les étapes est disponible sur github.
Cet article suppose des connaissances de base en data science et en Python.
La vision par ordinateur est une branche fascinante de l'apprentissage automatique qui s'est rapidement développée ces dernières années. Grâce à l'accès public à de nombreuses bibliothèques d'algorithmes(Tensorflow, OpenCv, Pytorch, Fastai...) ainsi qu'à des ensembles de données libres(CIFAR -10, Imagenet, IMDB...), il est très facile pour un scientifique ( data ) d'acquérir une expérience pratique dans ce domaine.
Mais saviez-vous que vous pouviez également accéder à des images satellites en libre accès ? Ces types d'images peuvent être utilisés dans de nombreux cas (agriculture, logistique, énergie...). Cependant, elles représentent des défis supplémentaires pour un scientifique de data en raison de leur taille et de leur complexité. J'ai écrit cet article pour partager certains de mes apprentissages clés en travaillant avec eux.
Étape 1 - Sélectionner la source ouverte du satellite data
Vous devez d'abord choisir le satellite que vous souhaitez utiliser. Les principales caractéristiques à prendre en compte sont les suivantes :
Voici les satellites dont le site data est disponible gratuitement :
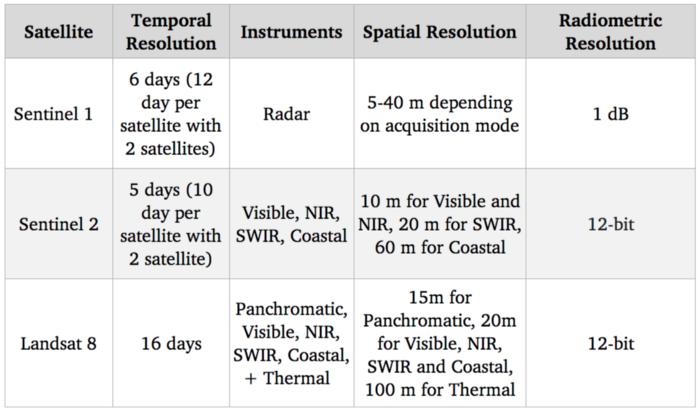
Benchmark des systèmes satellitaires
Notre objectif étant de détecter et d'identifier les cultures agricoles, le spectre visible (qui est utilisé pour fournir des images couleur standard) devrait être très utile. En outre, il existe des corrélations entre le niveau d'azote des cultures et les bandes spectrales NIR (proche infrarouge). En ce qui concerne les options gratuites, Sentinel 2 est actuellement le meilleur en termes de résolution avec 10 m pour ces bandes spectrales, nous allons donc utiliser ce satellite.
Bien entendu, une résolution de 10 m est bien inférieure à l'état de l'art en matière d'imagerie satellitaire, les satellites commerciaux atteignant une résolution de 30 cm. L'accès au satellite commercial data peut débloquer de nombreux cas d'utilisation, mais malgré une concurrence accrue avec l'arrivée de nouveaux acteurs sur ce marché, les prix restent élevés et donc difficiles à intégrer dans un cas d'utilisation à grande échelle.
Étape 2 - Collecte de l'imagerie satellite data à partir de Sentinel 2
Maintenant que nous avons sélectionné notre source satellitaire, la nouvelle étape consiste à télécharger les images. Sentinel 2 fait partie du programme Copernicus de l'Agence spatiale européenne. Nous pouvons accéder à son site data en utilisant l'API python SentinelSat.
Il faut d'abord créer un compte sur le Copernicus Open Access Hub. Notez que ce site web permet également d'interroger les images à l'aide d'une interface graphique.
Une façon de spécifier les zones que vous souhaitez explorer est de fournir à l'API un fichier geojson, qui est un json contenant les coordonnées GPS de la position (latitude et longitude) d'une zone. Les fichiers geojson peuvent être rapidement créés sur ce site web.
Vous pouvez ainsi interroger toutes les images satellites qui recoupent la zone indiquée.
Nous dressons ici la liste de toutes les images disponibles sur un site pour une zone et une période données :
api = SentinelAPI(
credentials["nom d'utilisateur"],
credentials["password"],
"https://scihub.copernicus.eu/dhus"
)
shape = geojson_to_wkt(read_geojson(geojson_path))
images = api.query(
shape,
date=(date(2020, 5, 1), date(2020, 5, 10)),
nom de la plateforme="Sentinel-2″,
niveau de traitement = "Level-2A",
pourcentage de couverture nuageuse=(0, 30)
)
images_df = api.to_dataframe(images)
Notez l'utilisation d'arguments :
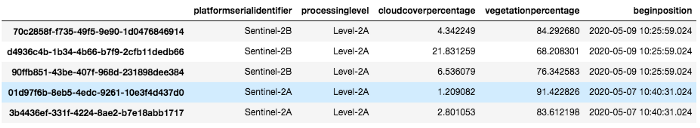
images_df
Images_df est un cadre de données pandas contenant toutes les images correspondant à notre requête. Nous en sélectionnons une avec une faible couverture cloud et la téléchargeons à l'aide de l'api :
api.download(uuid)
Étape 3 - Comprendre quelles bandes spectrales utiliser et gérer la quantification
Une fois le fichier zip téléchargé, nous pouvons constater qu'il contient en fait plusieurs images.
En effet, comme nous l'avons dit à l'étape 1, Sentinel 2 capture de nombreuses bandes spectrales, et pas seulement le rouge, le vert et le bleu, et chaque bande est capturée dans une seule image.
De plus, ses instruments n'ont pas tous la même résolution spatiale, il y a donc un sous-dossier pour chaque résolution différente 10, 20 et 60 mètres.
If we check the GRANULE/<image_id>/IMG_DATA folder we see that there is a subfolder for each different resolution: 10, 20 and 60 meters as the spectral bands. We are going to use those with max resolution 10m as they provide Band 2, 3, 4 and 8 which are Blue, Green, Red and NIR. Band 7, 8, 9 which are vegetation red edge (spectral bands between red and NIR that mark a transition in reflectance value for vegetation) could also be interesting for detecting crops but since they are lower resolution we are going to leave them aside now
Ici, nous chargeons chaque bande sous la forme d'une matrice numpy 2D :
def get_band(image_folder, band, resolution=10):
subfolder = [f for f in os.listdir(image_folder + “/GRANULE”) if f[0] == “L”][0]
image_folder_path = f”{image_folder}/GRANULE/{subfolder}/IMG_DATA/R{resolution}m”
image_files = [im for im in os.listdir(image_folder_path) if im[-4:] == “.jp2”]
selected_file = [im for im in image_files if im.split(“_”)[2] == band][0]
with rasterio.open(f”{image_folder_path}/{selected_file}”) as infile:
img = infile.read(1)
retour img
band_dict = {}
pour bande dans ["B02", "B03", "B04", "B08"] :
band_dict[band] = get_band(image_folder, band, 10)
Un test rapide pour s'assurer que tout fonctionne est d'essayer de recréer l'image en vraies couleurs (c'est-à-dire RGB) et de l'afficher en utilisant opencv et matplotlib :
img = cv2.merge((band_dict["B04"], band_dict["B03"], band_dict["B02"]))
plt.imshow(img)
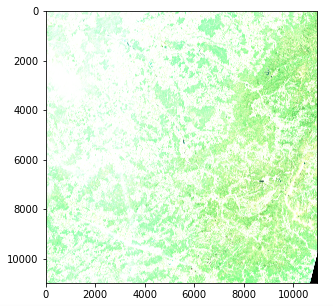
Image satellite - Problème d'intensité des couleurs (Copernicus Sentinel data 2020)
Cependant, il y a un problème avec l'intensité des couleurs. En effet, la quantification (nombre de valeurs possibles) diffère de la norme de matplotlib qui suppose soit une valeur int de 0-255, soit une valeur float de 0-1. Les images standard utilisent souvent une quantification de 8 bits (256 valeurs possibles), mais les images Sentinel 2 utilisent une quantification de 12 bits et un retraitement convertit les valeurs en un entier de 16 bits (65536 valeurs).
Si nous voulons convertir un nombre entier de 16 bits en un nombre entier de 8 bits, nous devrions en théorie diviser par 256, mais cela donne en fait une image très sombre puisque toute la gamme des valeurs possibles n'est pas utilisée. La valeur maximale de notre image est en fait de 16752 sur 65536 et peu de pixels atteignent en fait des valeurs supérieures à 4000, de sorte que la division par 8 donne en fait une image avec un contraste décent.
img_processed = img / 8
img_processed = img_processed.astype(int)
plt.imshow(img_processed)
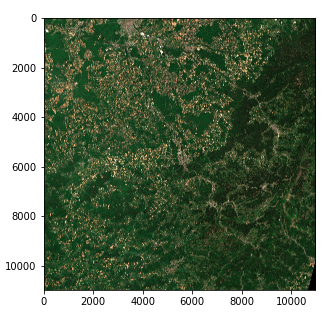
Image satellite - Intensité de la couleur droite (Copernicus Sentinel data 2020)
Nous avons maintenant un tenseur à 3 dimensions de forme (10980, 10980, 3) d'entiers à 8 bits sous la forme d'un tableau numpy.
On notera que :
Étape 4 - Diviser les images en tailles adaptées à l'apprentissage automatique
Nous pourrions essayer d'appliquer un algorithme à notre image actuelle, mais en pratique, cela ne fonctionnerait pas avec la plupart des techniques en raison de sa taille. Même avec une grande puissance de calcul, la plupart des algorithmes (et en particulier l'apprentissage profond) ne pourraient pas être appliqués.
Nous devons donc diviser l'image en fragments. Une question se pose : comment définir la taille des fragments ?
Nous décidons de diviser chaque image en une grille de 45 * 45 (45 étant commodément un diviseur de 10980).
Passé ce paramétrage, cette étape est directe et utilise la manipulation de tableaux numpy. Nous stockons nos valeurs dans un dict en utilisant les tuples (x, y) comme clés.
frag_count = 45
frag_size = int(img_processed.shape[0] / frag_count)
frag_dict = {}
pour y, x dans itertools.product(range(frag_count), range(frag_count)) :
frag_dict[(x, y)] = img_processed[y*frag_size : (y+1)*frag_size,
x*taille_frag : (x+1)*taille_frag, :]
plt.imshow(frag_dict[(10, 10)])
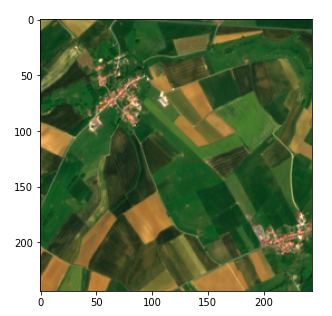
Fragment d'image satellite (Copernicus Sentinel data 2020)
Étape 5 - Reliez votre site data aux images satellite en utilisant la conversion GPS/UTM

Enfin, nous devons relier nos images au site data que nous avons sur les champs agricoles pour permettre une approche d'apprentissage automatique supervisé. Dans notre cas, nous disposons de data sur les champs agricoles sous la forme d'une liste de coordonnées GPS (latitude, longitude), similaire au geojson que nous avons utilisé à l'étape 2, et nous voulons être en mesure de trouver la position de leurs pixels. Cependant, par souci de clarté, je commencerai par présenter une conversion de pixels en coordonnées GPS, puis je montrerai l'inverse.
Les métadonnées collectées lors de la demande de l'API SentinelSat fournissent les coordonnées GPS des coins de l'image (colonne footprint) et nous voulons les coordonnées d'un pixel spécifique. La latitude et la longitude, qui sont des valeurs angulaires, n'évoluent pas linéairement au sein d'une image, de sorte qu'il n'est pas possible d'effectuer un simple rapport en utilisant la position du pixel. Il existe des moyens de résoudre ce problème à l'aide d'équations mathématiques, mais des solutions prêtes à l'emploi existent déjà en Python.
Une solution rapide consiste à convertir la position du pixel en UTM (Universal Transverse Mercator) dans lequel une position est définie par une zone ainsi qu'une position (x, y) (en unité de mètre) qui évolue linéairement avec l'image. Nous pouvons alors utiliser une conversion UTM vers Latitude / Longitude fournie dans la bibliothèque utm. Pour ce faire, nous devons d'abord obtenir la zone UTM et la position du coin supérieur gauche de l'image, qui peuvent être trouvées dans les métadonnées de l'image couleur.
#Obtention des données de viande
transform = rasterio.open(tci_file_path, driver='JP2OpenJPEG').transform
zone_number = int(tci_file_path.split("/")[-1][1:3])
zone_letter = tci_file_path.split("/")[-1][0]
utm_x, utm_y = transform[2], transform[5]
# Conversion de la position du pixel en utm
east = utm_x + pixel_column * 10
north = utm_y + pixel_row * - 10
# Convertir UTM en latitude et longitude
latitude, longitude = utm.to_latlon(east, north, zone_number, zone_letter)
La conversion de la position GPS en pixels s'effectue à l'aide des formules inverses. Dans ce cas, nous devons nous assurer que la zone obtenue correspond à notre image. Cela peut être fait en spécifiant la zone. Si notre objet n'est pas présent dans l'image, il en résultera une valeur de pixel hors limites.
# Conversion de la latitude et de la longitude en UTM
east, north, zone_number, zone_letter = utm.from_latlon(
latitude, longitude, force_zone_number=zone_number
)
# Conversion de l'UTM en colonne et en ligne
pixe_column = round((east - utm_x) / 10)
pixel_row = round((north - utm_y) / -10)
voir les données brutes
Conclusion
Nous sommes maintenant prêts à utiliser les images satellites pour l'apprentissage automatique !
Dans le prochain article de cette série en deux parties, nous verrons comment nous pouvons utiliser ces images pour détecter et classer les surfaces des champs en utilisant l'apprentissage automatique supervisé et non supervisé.
Merci de votre lecture et n'hésitez pas à suivre le blog Artefact tech si vous souhaitez être averti de la sortie de ce prochain article !

 BLOG
BLOG